 Ανάλυση γονιδιακής εμπλουτισμού που είναι μία από τις πολλές προσεγγίσεις για τη ανάλυση της γονιδιακής έκφρασης δεδομένα προφίλ και περιγράφεται στο α χαρτίαπό τους εργαζόμενους στο Ινστιτούτο Broad.
Ανάλυση γονιδιακής εμπλουτισμού που είναι μία από τις πολλές προσεγγίσεις για τη ανάλυση της γονιδιακής έκφρασης δεδομένα προφίλ και περιγράφεται στο α χαρτίαπό τους εργαζόμενους στο Ινστιτούτο Broad.
Η βασική ιδέα είχε ζητηθεί από την παρατήρηση ότι η μελέτη μεμονωμένα γονίδια που δείχνει την πιο σημαντική διαφορά στο επίπεδο έκφρασης μεταξύ των δύο κρατών ή φαινοτύπου είναι λείπει σε μηχανιστική εικόνα. Αντί, είναι πιο λογικό να λάβει ένα σύνολο γονιδίων και συμφωνεί με ορισμένες βιολογικού δεσμού, και να ζητήσει από την ερώτηση - δεν το σύνολο δείχνουν κάθε στατιστικώς σημαντικό εμπλουτισμό σε αυτά τα γονίδια που έχουν διαφορική έκφραση?
Α σετ γονιδίων μπορεί να επιλεγεί, a priori, για διάφορους λόγους π.χ.. το σύνολο των γονιδίων που είναι γνωστό ότι επηρεάζεται από πάνω- ή υπό-έκφραση ενός μικρο-RNA, ή ίσως ένα σύνολο επιλέγονται με βάση την χρωμοσωμική τοποθεσία, ή γονίδια για τα οποία μοριακή λειτουργία, κυτταρική συνιστώσα και / ή βιολογική διεργασία έχουν ανατεθεί χρησιμοποιώντας τα ελεγχόμενα λεξιλόγια της Οντολογία γονιδίων.
Ένα πλεονέκτημα της προσέγγισης GSEA είναι ότι είναι δυνατόν να ενσωματωθούν σας πλήρη στοιχεία που, δεν είναι μόνο οι μεταγραφές με ένα αυθαίρετα επιλεγμένο όριο διαφορική έκφραση. Είμαι βέβαιος ότι πολλοί άνθρωποι ανάγνωση αυτού θα πρέπει να σκέφτεται - «Πώς μπορεί να είναι ΟΚ για να χρησιμοποιήσετε το πλήρες σύνολο δεδομένων? Κανονικά θα εξετάσει μόνο τα γονίδια με >2 (Ή άλλα αγαπημένα αξία)-πλάσια διαφορική έκφραση. "Ο λόγος η προσέγγιση είναι έγκυρη είναι ότι τα γονίδια που εκφράζεται σε χαμηλά επίπεδα ή με μεγάλη διακύμανση μεταξύ των επαναλήψεων δεν συμβάλλουν στην κύρια μετρική χρησιμοποιείται από GSEA, το «Αποτελέσματα εμπλουτισμού» (ES).
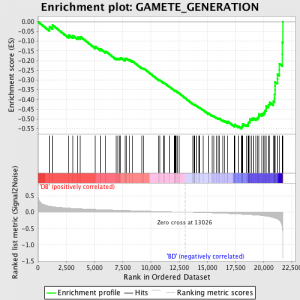
GSEA λειτουργεί από την πρώτη κατάταξη η αξία έκφρασης για κάθε γονίδιο από σήματος προς θόρυβο αναλογία - υπολογισμό της διαφοράς μεταξύ των μέσων τιμών για τα δείγματα που αντιπροσωπεύουν κάθε φαινότυπο και κλιμάκωση αυτούς από το άθροισμα των τυπικών αποκλίσεων. Αυτό σημαίνει ότι τα γονίδια με μεγάλες διαφορές σε επίπεδο έκφρασης μεταξύ των διαφόρων κρατών και μικρή διακύμανση μεταξύ των βιολογικών επαναλήψεων κατατάσσονται σε μεγάλο βαθμό.
Το επόμενο βήμα είναι ότι το ES, το κύριο στατιστικό στοιχείο που παράγεται από GSEA, υπολογίζεται για κάθε σετ γονιδίων - στην GSEA εγχειρίδιο, το οποίο τεκμηριώνει το λογισμικό άριστα, αναφέρει:
"Όλα τα γονίδια πρώτα ανάλογα με το σήμα τους προς θόρυβο, τότε η ES υπολογίζεται με "πόδια" κάτω από τον πίνακα κατάταξης των γονιδίων αύξηση ένα στρώσιμο ποσό στατιστική όταν ένα γονίδιο είναι στο σύνολο γονιδίων και μειώνεται όταν δεν είναι. Ο μέγεθος της προσαύξησης εξαρτάται από τη συσχέτιση του γονιδίου με ένα φαινότυπο. Η ES είναι η μέγιστη απόκλιση από το μηδέν συνάντησε στο περπάτημα τη λίστα. Α θετικός ES δείχνει γονίδιο εμπλουτισμού καθοριστεί στο κορυφή του πίνακα κατάταξης; ένα αρνητικός ES δείχνει γονίδιο εμπλουτισμού καθοριστεί στο κάτω μέρος του πίνακα κατάταξης. "
Οι τιμές είναι ES κανονικοποιείται με βάση το μέγεθος σετ γονιδίων και στη συνέχεια ένα ψευδείς ποσοστό ανακάλυψη υπολογίζεται, για να δώσει μια εκτιμώμενη πιθανότητα ψευδώς θετικά. GSEA χρησιμοποιεί μια πολύ χαλαρή προκαθορισμένη τιμή 25%, το οποίο είναι κατάλληλο για τη δημιουργία υποθέσεων με ένα σχετικά μεγάλο αριθμό επαναλήψεων των βιολογικών.
Οι επιστήμονες που εργάζονται σε δεδομένα από μη ανθρώπινο δείγματα μπορούν να εξακολουθούν να χρησιμοποιούν GSEA, αλλά πρέπει ναΠροσοχή - Η σύμβολα γονιδίων χρησιμοποιείται από GSEA είναι "μετάφραση"Από την ανθρώπινη δηλαδή τα ισοδύναμά τους. Τα αναγνωριστικά που χρησιμοποιούνται για τα γονίδια από τα είδη που σας ενδιαφέρουν εκπροσωπούνται στη μικροσυστοιχία μετατρέπονται σε σύμβολα για τους ανθρώπινων orthologues, συνέχεια χρησιμοποιήθηκε στην ανάλυση. Subramanian και οι συνεργάτες του αξίωση ότι αυτή η μετατροπή έχει μικρή ή καμία επίδραση σχετικά με τη χρησιμότητα της GSEA; έχει χρησιμοποιηθεί με επιτυχία σε πολλαπλές μη ανθρώπινο είδος, αλλά φυσικά αυτό θα πρέπει να ληφθεί υπόψη κατά τη διερεύνηση αποτελέσματα αναλυτικά.
Για μια εξαιρετική, σε βάθος, επανεξέταση των εργαλείων οδού, συμβουλευτείτε:
Μια άλλη καλή πηγή των συμβουλών σχετικά με την ανάλυση οδό, ειδικά για όσους είναι εξοικειωμένοι με το πακέτο R είναι στατιστικά εδώ.
Περαιτέρω ανάγνωση