 Gene Set Enrichment analyysi on yksi monista tavoista analyysi geenien profiilitietoja ja se on kuvattu paperialkaen työntekijöiden Broad Instituutissa.
Gene Set Enrichment analyysi on yksi monista tavoista analyysi geenien profiilitietoja ja se on kuvattu paperialkaen työntekijöiden Broad Instituutissa.
Perusajatuksena syynä oli havainto, että opiskelu yksittäiset geenit jossa merkittävin ero ilmaisun tasolla kahden valtioiden tai fenotyyppi on puuttuu mekanistinen käsitys. Sen sijaan, järkevämpää ottaa joukko geenejä jakaa joitakin biologiset linkki, ja kysyä - ei koko joukko osoittaa tilastollisesti kartutti merkittävästi niissä geenit, jotka on ero ilmaisun?
A geeniperimä voidaan valita, priori, useista syistä esim.. joukko geenejä tiedetään vaikuttavan yli- tai-ilmaisua mikro-RNA, tai ehkä asettaa valitaan perustuu sijainti kromosomissa, tai geenejä, joiden molekyyli-toiminto, solukomponentista ja / tai biologinen prosessi on valittu käyttäen ohjattu sanastoja Gene ontologia.
Yksi etu GSEA lähestymistapa on se, että on mahdollista sisällyttää oman täydelliset tiedot asetetaan, ei vain niille, selostukset valittu mielivaltaisesti ero ilmaisun kynnys. Olen varma, että monet ihmiset lukevat tätä ajatella - "Miten voi olla hyväksyttävää käyttää täydellinen aineisto? Normaalisti olisin vain pitää geenejä >2 (Tai muita suosikki arvo)-kertainen ero ilmaisua. "Syy lähestymistapa on voimassa, että ekspressoidut alhainen tai suuri varianssi toistojen välinen eivät edistä tärkein metristä käyttää GSEA, "rikastamiseen pisteet" (ES).
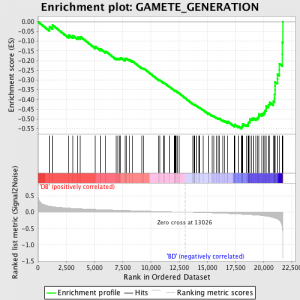
GSEA toimii ensin sijoitus lausekkeen arvo kunkin geenin Häiriöetäisyys suhde - lasketaan ero keskiarvot näytteitä, jotka edustavat kunkin fenotyyppi ja niiden laajentamista summalla keskihajonnat. Tämä tarkoittaa sitä, että geenit suuria eroja ilmaisun tasolla eri valtioiden ja vähän vaihtelua biologisen rinnakkaista sijoittuu hyvin.
Seuraava vaihe on, että ES, Ensisijainen tilastotieto syntyy GSEA, lasketaan kunkin geenin set - in GSEA käsikirja, joka dokumentoi ohjelmisto erinomaisesti, todetaan:
"Kaikki geenit asetetaan paremmuusjärjestykseen niiden signaali-kohina-suhde, Sitten ES lasketaan "kävely" alas paremmuusjärjestykseen geenien lisää a sisäänajon summa tilastotieto kun geeni on geeni asettaa ja laskussa kun se ei ole. The suuruus ulkoreunasta riippuu korrelaatio geenin kanssa fenotyyppi. ES on suurin poikkeama nollasta kohdataan kävely lista. A positiivinen ES osoittaa geeniperimä rikastumiseen toppi ja paremmuusjärjestykseen; a negatiivinen ES osoittaa geeniperimä rikastumiseen pohja ja paremmuusjärjestykseen. "
ES-arvot ovat normalisoitui perustuu geeniperimä kokoa ja sitten vääriä löytö korko lasketaan, antaa arvioitu todennäköisyys vääriä positiivisia. GSEA käyttää hyvin rento oletusarvo 25%, joka on sopiva hypoteesi sukupolven suhteellisen suuri määrä biologisia rinnakkaisnäytteiden.
Tutkijat työskentelevät tietoja ei-ihmis- näytteitä voidaan silti käyttää GSEA, mutta täytyyvarokaa - geeni symbolit käyttämät GSEA ovat "käännetty"Niiden ihmisten vastineet so. tunnisteita käytetään geenien lajisi kohteisiin edustettuna mikrosirujen muunnetaan symboleiksi ihmisen orthologues, Sitten käytetään analyysissa. Subramanian ja työtovereiden vaatia että tämä muuntaminen on vain vähän tai ei vaikutusta siitä, olisiko GSEA; sitä on käytetty menestyksekkäästi useiden ei-ihmislajin, mutta tietenkin tämä on pidettävä mielessä tutkittaessa tuloksia yksityiskohtaisesti.
Erinomaisesta, syvällinen, tarkastelun polku työkalut, konsultoida:
Toinen hyvä lähde neuvoja polku analyysi, erityisesti niille tuttuja R tilastojen paketti on täällä.
Kirjallisuutta

Yksi paluuviite
[...] tietojen genomin laajuinen differentiaaliseksi-ilmaisi selostukset. Käyttämällä Broad Instituutissa ihmeellinen GSEA työkalu, Olen arvioinut tilastollista näyttöä siitä, että tietyn geenin ontologia ehdot ja reitit olivat [...]