 基因集富集分析的许多方法之一 分析基因表达 描述文件数据,并在所述 纸工人在Broad研究院.
基因集富集分析的许多方法之一 分析基因表达 描述文件数据,并在所述 纸工人在Broad研究院.
提示通过观察,学习的基本概念 单个基因 显示两种状态之间或表型表达水平最显着的差异是 缺乏机械洞察力. 代替, 采取更有意义 基因组 分享一些 生物链接, 问的问题 - 不显示任何统计学整套 显着富集 在那些有差异表达的基因?
一 基因组 可以选择, 先验, 一些原因,例如. 由以上的影响已知的基因集- 或根据一个微RNA的表达, 或者所选择的一组的基础上的染色体位置, 或基因的分子功能, 细胞成分和 / 或生物过程已分配使用的受控词表 基因本体论.
GSEA方法的优点之一是,它是可以掺入的 完整的数据集, 不只是那些成绩单和一个任意选定的差异表达门槛. 我敢肯定,很多人阅读本思考 - “这怎么可能确定使用完整的数据集? 通常情况下,我只会考虑基因 >2 (或其他喜爱的价值)-倍差异表达。“原因的方法是有效的,是在较低水平或复制较大差异表达的基因不利于GSEA使用的主要指标, '富集得分“ (ES).
GSEA第一 排行 每个基因的表达值 信号噪声 比例 - 计算代表每个表型的样品的平均值之间的差异和缩放它们的标准偏差的总和. 这意味着,基因表达水平差异较大的不同状态之间和生物之间的变化不大复制的排名高度.
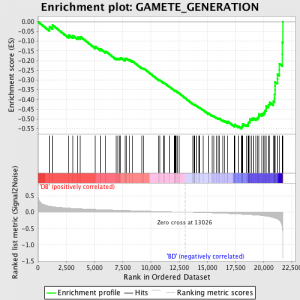
下一个步骤是对ES, GSEA所产生的主要统计数据, 计算每个基因的集 - GSEA手册, 出色记录软件, 它指出:
“所有基因的信号噪声比排名第一, 然后在ES的计算方法是“走”下来的排名列表基因 增加 一 运行总和 统计时,一个基因的基因组中,并 减少 它时,它不是. 该 大小 的增量取决于 相关 与基因 型. ES是从零走在列表中遇到的最大偏差. 一 积极 ES表明基因组富集 顶部 位列榜单; 一 负 ES表明基因组富集 底部 位列榜单。“
ES值 归 基于基因组的大小,然后一个 错误发现率 计算, 误报的概率的估计. GSEA使用一个非常宽松的默认值 25%, 这是适于生成假设有相对大量的生物复制.
数据科学家 non-human 样品仍然可以使用GSEA, 但需要提防 - 在 基因符号 使用GSEA“翻译“从他们的人力等值i.e. 标识符用于物种基因芯片为代表的利息转换成他们的符号 人类同源基因, 然后在分析中使用. Subramanian和他的同事们 声称 这种转换具有很少或 没有效果 GSEA的效用; 它已成功地用于在多个非人类物种, 但当然,这必须牢记的详细调查结果时,.
对于一个优秀的, 在深入, 审查通路工具, 请教:
另一个很好的来源途径分析建议, 尤其是那些熟悉的R统计软件包 这里.
延伸阅读

一 引用
[...] 数据的全基因组差异表达的转录. 使用Broad研究所的奇妙的GSEA工具, 我评估的统计证据表明,特定的基因本体条款和途径 [...]