 Gene Análise Enriquecimento Set é uma das muitas abordagens, o A análise da expressão do gene dados do perfil e é descrita em um papeldos trabalhadores do Instituto Broad.
Gene Análise Enriquecimento Set é uma das muitas abordagens, o A análise da expressão do gene dados do perfil e é descrita em um papeldos trabalhadores do Instituto Broad.
O conceito básico foi motivada pela constatação de que estudar genes individuais mostrando a diferença mais significativa nos níveis de expressão entre dois estados ou fenótipos é falta de uma visão mecanicista. Em vez, faz mais sentido ter um conjunto de genes partilha de alguns vínculo biológico, e fazer a pergunta - se o conjunto mostrar qualquer estatisticamente enriquecimento significativo nesses genes que têm expressão diferencial?
A set gene pode ser escolhido, a priori, por uma série de razões e.g.. o conjunto de genes que se sabe ser influenciada por mais de- ou sob-expressão de um micro-RNA, ou, talvez, um conjunto escolhido com base na localização cromossómica, ou genes para os quais a função molecular, componente celular e / ou processo biológico foram atribuídos utilizando os vocabulários controlados do Gene Ontology.
Uma vantagem da abordagem GSEA é que é possível incorporar o dados completos definir, não apenas os transcritos com um limiar escolhido arbitrariamente expressão diferencial. Tenho certeza de que muitas pessoas que lêem este estará pensando - "Como pode ser OK para usar o conjunto de dados completo? Normalmente eu consideraria somente genes com >2 (OU valor outro favorito)-a expressão diferencial de dobragem. "A razão, a abordagem é válida é que os genes expressos em níveis baixos ou com grande variação entre repetições não contribuem para a métrica principal utilizado pelo GSEA, o 'pontuação enriquecimento' (ES).
AGEE funciona através da posição o valor da expressão de cada gene por sinalizar ao ruído ratio - cálculo da diferença entre os valores médios para as amostras representativas de cada fenótipo e escalonando-los pela soma dos desvios padrão. Isto significa que genes com grandes diferenças no nível de expressão entre diferentes estados e pouca variação entre réplicas biológicas são classificados altamente.
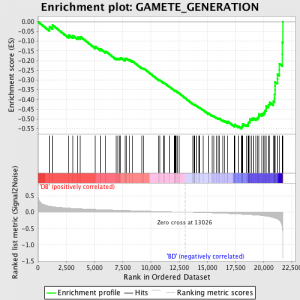
O próximo passo é a de que o ES, a estatística primária gerada pelo GSEA, é calculada para cada conjunto de genes - no manual GSEA, que documenta o software excelente, afirma:
"Todos os genes são inicialmente classificados pela sua relação sinal-ruído, em seguida, o ES é calculado por "andar" para baixo da lista de classificação dos genes aumentar um execução soma- estatística quando um gene está no conjunto de genes e decrescente que, quando não é. O magnitude do incremento depende da correlação do gene com um fenótipo. O ES é o desvio máximo de zero encontrou uma curta lista. A positivo ES indica enriquecimento gene conjunto no topo da lista de classificação; um negativo ES indica enriquecimento gene conjunto no fundo da lista de classificação. "
Os valores são ES normalizada com base no tamanho do conjunto de genes e, em seguida, uma taxa de descoberta de falsa é calculado, para dar uma probabilidade estimada de falsos positivos. AGEE usa um valor padrão muito relaxado 25%, que é adequado para a geração de hipóteses com um número relativamente grande de réplicas biológicas.
Os cientistas que trabalham em dados a partir de não humano amostras ainda pode usar AGEE, mas precisater cuidado - O símbolos de genes utilizado pelo GSEA são "traduzidos"Do seu i.e. equivalentes de humano. identificadores utilizados por genes de suas espécies de interesse representado no microarray são convertidas em símbolos para a sua ortólogos humanos, em seguida, utilizada na análise. Subramanian e colegas reivindicar que esta conversão tem pouco ou nenhum efeito sobre a utilidade dos GSEA; tem sido utilizada com sucesso em várias espécies não humanas, mas é claro que isso deve ser mantido em mente quando se investiga resultados em detalhes.
Para uma excelente, em profundidade, revisão dos instrumentos via, consultar:
Outra boa fonte de conselhos sobre análise de percurso, especialmente para aqueles familiarizados com o pacote estatístico R é aqui.
Outras leituras

Um Trackback
[...] data genome-wide for differentially-expressed transcripts. Using the Broad Institute’s marvellous GSEA tool, I assessed the statistical evidence that specific Gene Ontology terms and pathways were [...]