 Analysis Set Gene arricchimento è uno dei molti approcci al analisi di espressione genica dati del profilo ed è descritto in un cartada parte dei lavoratori durante il Broad Institute.
Analysis Set Gene arricchimento è uno dei molti approcci al analisi di espressione genica dati del profilo ed è descritto in un cartada parte dei lavoratori durante il Broad Institute.
Il concetto di base è stato richiesto dalla constatazione che lo studio singoli geni mostra la differenza più significativa nel livello di espressione tra due Stati o fenotipi è privo di comprensione meccanicistica. Invece, ha più senso di prendere una set di geni la condivisione di alcuni legame biologico, e la domanda - è l'insieme di tutto mostra alcuna statisticamente arricchimento significativo in quei geni che hanno un'espressione differenziale?
La gene set può essere scelto, a priori, per una serie di ragioni e.g. l'insieme di geni noti per essere influenzato da oltre- o sotto-espressione di un micro-RNA, o forse un gruppo scelto in base alla posizione cromosomica, o geni per i quali la funzione molecolare, componente cellulare e / o biologico sono stati assegnati utilizzando i vocabolari controllati della Gene Ontology.
Un vantaggio dell'approccio GSEA è che è possibile incorporare tua dati completi set, non solo quelli trascritti con una soglia differenziale scelto arbitrariamente espressione. Sono sicuro che molte persone che leggono questo sarà il pensiero - "Come può essere OK per utilizzare il set di dati completo? Normalmente avrei in considerazione solo i geni con >2 (O altro valore preferito)-espressione differenziale piega. "La ragione per cui l'approccio è valido è che i geni espressi a livelli bassi o con grande varianza tra replicati non contribuiscono alla metrica principale utilizzato da GSEA, la 'arricchimento punteggio' (ES).
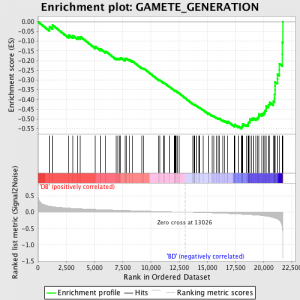
GSEA funziona anzitutto mediante la posizione il valore di espressione per ciascun gene da segnale-rumore rapporto - calcolando la differenza tra i valori medi per i campioni rappresentano ciascun fenotipo e ridimensionandoli dalla somma delle deviazioni standard. Ciò significa che i geni con grandi differenze nel livello di espressione tra i diversi stati e variazione minima tra replicati biologici sono classificati molto.
Il passo successivo è che l'ES, la statistica primario, GSEA, sono calcolati per ciascun gene - nel manuale GSEA, che documenta il software eccellente, si afferma:
"Tutti i geni vengono prima ordinati per il loro rapporto segnale-rumore, allora la ES è calcolata "camminare" lungo la graduatoria dei geni crescente un running-sum statistica quando un gene è nel set gene e decrescente quando non è. Il magnitudine dell'incremento dipende correlazione del gene con una fenotipo. L'ES è la massima deviazione da zero incontrato in piedi la lista. La positivo ES indica set arricchimento genica a top della graduatoria; un negativo ES indica set arricchimento genica a fondo della graduatoria. "
I valori sono ES normalizzato in base alle dimensioni del gene e poi un set scoperta di falsi è calcolato, dare una probabilità stimata di falsi positivi. GSEA utilizza un valore di default molto rilassato di 25%, che è adatto per la generazione di ipotesi con un numero relativamente grande di biologico replicati.
Scienziati che lavorano su dati da non umano i campioni possono ancora usare GSEA, ma è necessariodiffidare - Il gene simboli utilizzato da GSEA sono "tradotto"Dal loro i.e umana equivalenti. identificatori utilizzati per i geni dei tuoi specie di interesse rappresentate nel microarray vengono convertiti in simboli per la loro ortologhi umani, poi utilizzato nell'analisi. Subramanian e colleghi rivendicazione che questa conversione ha poco o nessun effetto sull'utilità di GSEA; è stato usato con successo in molteplici specie non umana, ma naturalmente questo deve essere tenuto presente nell'ambito delle indagini risultati in dettaglio.
Per un eccellente, approfondita, revisione degli strumenti pathway, consultare:
Un'altra buona fonte di consigli su analisi di percorso, soprattutto per chi ha familiarità con il pacchetto R statistiche è qui.
Ulteriori letture

Un Trackback
[...] dati genome-wide per trascritti differenzialmente espressi. Utilizzando meraviglioso strumento GSEA il Broad Institute, Ho valutato l'evidenza statistica che specifici termini Gene Ontology e percorsi erano [...]