 Gene Set Enrichment Analysis is een van de vele benaderingen analyse van genexpressie profielgegevens en wordt beschreven in een papiervan de werknemers op het Broad Institute.
Gene Set Enrichment Analysis is een van de vele benaderingen analyse van genexpressie profielgegevens en wordt beschreven in een papiervan de werknemers op het Broad Institute.
Het basisconcept is ingegeven door de observatie dat het bestuderen individuele genen toont de meest significante verschil in expressie niveau tussen twee staten of fenotypes is ontbreekt in mechanistisch inzicht. In plaats daarvan, is het zinvoller om een te nemen set van genen delen van een aantal biologische band, en de vraag - niet de hele set tonen enkel statistisch aanzienlijke verrijking in die genen die differentiële expressie hebben?
Een genreeks worden gekozen, a priori, voor een aantal redenen b.v.. de reeks genen bekend te worden beïnvloed door via- of onder-expressie van een micro-RNA, of misschien een set gekozen op basis van chromosomale locatie, of genen waarvoor moleculaire functie, cellulaire component en / of biologisch proces zijn toegewezen met behulp van de gecontroleerde woordenlijsten van de Ontology van het gen.
Een voordeel van de GSEA benadering is dat het mogelijk is te nemen uw set volledige gegevens, niet alleen die transcripten met een willekeurig gekozen differentiële expressie drempel. Ik ben er zeker van dat veel mensen die dit lezen zullen denken - "Hoe kan het OK om de volledige dataset te gebruiken? Normaal gesproken zou ik alleen genen overwegen met >2 (OF andere favoriet waarde)-voudige differentiële expressie. "De reden de benadering geldt dat genen die op een laag niveau of grote verschil tussen herhaalde niet bijdragen tot de belangrijkste metrische gebruikt GSEA, de 'verrijking score' (ES).
GSEA werkt door eerst ranking de uitdrukking waarde voor elk gen door signaal aan lawaai verhouding - berekenen van het verschil tussen de gemiddelde waarden van monsters die elk fenotype te schalen door de som van de standaardafwijkingen. Dit betekent dat de genen met grote verschillen in expressie niveau tussen de verschillende staten en weinig variatie tussen biologische repliceert zijn zeer gerangschikt.
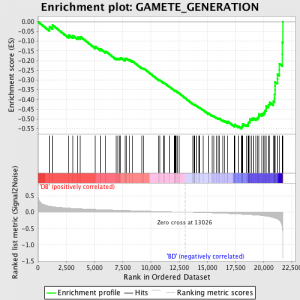
De volgende stap is dat de ES, de primaire statistiek gegenereerd door GSEA, berekend voor elk gen set - in de handleiding GSEA, waarop de software uitstekend documenteert, verklaart zij:
"Alle genen worden eerst in volgorde van hun signaal-ruisverhouding, dan is de ES wordt berekend door "lopen" onderaan de ranglijst van genen toenemende een running-sum Statistiek wanneer een gen is in het gen set en afnemende wanneer het niet. De omvang van de toename hangt af van de correlatie van het gen met een fenotype. De ES is de maximale afwijking van nul ondervonden bij het lopen van de lijst. Een positief ES geeft genreeks verrijking bij de top van de ranglijst; een negatief ES geeft genreeks verrijking bij de bodem van de ranglijst. "
De ES waarden genormaliseerd gebaseerd op genen setgrootte en vervolgens een valse ontdekking tarief berekend, een geschatte kans op valse positieven te geven. GSEA maakt gebruik van een zeer ontspannen standaardwaarde van 25%, die geschikt is voor hypothesen met een relatief groot aantal biologische repliceert.
Wetenschappers werken op gegevens van non-human monsters kunnen nog steeds gebruik maken GSEA, maar moetenoppassen - De gen symbolen gebruikt door GSEA zijn "vertaald"Van hun menselijke equivalenten i.e.. identifiers gebruikt voor genen van jullie soort van belang zijn vertegenwoordigd in de microarray worden omgezet in symbolen voor hun menselijke orthologa, vervolgens gebruikt in de analyse. Subramanian en collega's beweren dat deze omzetting weinig of geen effect het nut van GSEA; Het is met succes gebruikt in meerdere niet-menselijke species, maar natuurlijk dit in het achterhoofd moet worden gehouden bij het onderzoek naar de resultaten in detail.
Voor een uitstekende, diepgaande, herziening van pathway gereedschappen, raadplegen:
Een andere goede bron van advies voor pathway-analyse, vooral voor die bekend zijn met de R statistieken pakket is hier.
Verder lezen

Één Trackback
[...] gegevens genoom-brede voor-differentieel uitgedrukt transcripten. Behulp van prachtige GSEA instrument de globale Instituut, Ik het statistische bewijs dat specifieke Gene Ontology termen en paden waren geëvalueerd [...]