 Set Analysis Gene Wzbogacanie jest jednym z wielu podejść do Analiza ekspresji genów Dane profilowe i jest opisany w papierz pracowników w Broad Institute.
Set Analysis Gene Wzbogacanie jest jednym z wielu podejść do Analiza ekspresji genów Dane profilowe i jest opisany w papierz pracowników w Broad Institute.
Podstawowa koncepcja został poproszony przez obserwację, że studiuje Poszczególne geny pokazując najbardziej znaczącą różnicę w poziomie ekspresji pomiędzy dwoma stanami i fenotypy jest brakuje mechanistycznego wglądu. Zamiast, większy sens brać zestaw genów dzielenie niektórych Link biologiczna, i zadać sobie pytanie - czy cały zestaw wykazał statystycznie znaczne wzbogacenie w tych genów, które mają ekspresję różnicową?
A zestaw genów może być wybrana, apriorycznie, z kilku powodów e.g. zestaw genów znanych wpływać na- lub pod-wyraz mikro-RNA, a może zestaw wybierany na podstawie lokalizacji chromosomowej, lub geny, dla których funkcja cząsteczkowa, składnikiem komórkowym i / lub biologiczne zostały przypisane przy użyciu kontrolowanych słowników Ontologia Gene.
Jedną z zalet tej GSEA podejścia jest to, że jest możliwe, aby uwzględnić wprowadzone zestaw kompletnych danych, nie tylko te stenogramy z dowolnie wybranego progu ekspresji różnicowej. Jestem pewien, że wiele osób czyta ten będzie myślał - "Jak to może być OK, aby korzystać z pełnego zestawu danych? Normalnie bym pod uwagę tylko genów z >2 (Lub innego ulubionego wartość za)-fold wyrażenie różnica. "Powodem podejście jest ważne to, że geny wyrażone na niskim poziomie lub z dużym sprzeczności między powtórzeń nie przyczyniają się do głównego metryczny używany przez GSEA, 'wynik wzbogacanie' (ES).
GSEA działa przez pierwsze ranking Wartość ekspresji każdego genu przez stosunek sygnału do szumu Stosunek - obliczenie różnicy między średnimi wartościami dla próbek reprezentujących każdą fenotyp i zwiększenia ich przez sumę odchyleń standardowych. Oznacza to, że geny z dużych różnic w poziomie ekspresji pomiędzy różnymi państwami i niewielka zmienność między biologicznymi powtórzeń są wysoko w rankingu.
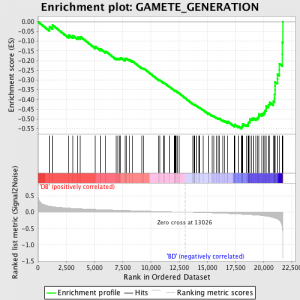
Następnym krokiem jest to, że ES, Podstawowym generowane przez GSEA statystyka, jest obliczana dla każdego zestawu genów - w GSEA instrukcji, dokumentująca oprogramowanie doskonale, stwierdza:
"Wszystkie geny są najpierw wg ich stosunku sygnału do szumu, następnie ES jest obliczana przez "chodzenie" w dół listy rankingowej genów wzrastający a running-sum Statystyka, gdy gen jest w zestaw genów i zmniejszenie to wtedy, gdy nie jest. The wielkość przyrostu zależy korelacja genu z fenotyp. ES jest maksymalne odchylenie od zera napotkał w chodzeniu listę. A pozytywny ES oznacza wzbogacenie zestawu genów w top z listy rankingowej; a negatywny ES oznacza wzbogacenie zestawu genów w dolny na liście rankingowej. "
Wartości ES są znormalizowane w oparciu o wielkości zestawu genów, a następnie fałszywych odkrycie jest obliczana, dać szacunkowe prawdopodobieństwo fałszywych alarmów. GSEA używa bardzo zrelaksowany wartości domyślnej 25%, która jest odpowiednia do wytwarzania hipotezy o stosunkowo dużej liczbie powtórzeń biologicznych.
Naukowcy pracujący na danych z non-human próbki mogą nadal korzystać GSEA, ale musząstrzec się - Symbole genów wykorzystywane przez GSEA są "przetłumaczony"Z ich ludzkiej i.e ekwiwalentów. identyfikatory używane do genów z waszego gatunku interesów reprezentowanych na mikromacierzy są konwertowane na symbole na ich ludzkie ortologów, następnie stosuje się do analizy. Subramanian i koledzy dochodzić że ta konwersja ma niewiele lub bez efektu na użyteczność GSEA; został on z powodzeniem stosowany w wielu gatunków innych niż człowiek, ale oczywiście muszą być brane pod uwagę podczas badania wyników szczegółowo.
Na znakomity, Dogłębna, Przegląd narzędzi szlaku, konsultować:
Innym dobrym źródłem informacji na analizie szlaku, zwłaszcza dla tych, którzy znają pakietu statystycznego R jest tutaj.
Chcesz wiedzieć więcej?

Jeden Trackback
[...] dane dla całego genomu transkryptów zróżnicowanej ekspresji. Korzystanie wspaniałe narzędzie Broad Institute w GSEA, I ocenić statystycznego dowodu, że konkretne terminy ontologii genów i ścieżki były [...]